13.1 Árboles de decisión
Los árboles de decisión son útiles para entender la estructura de un conjunto de datos. Sirven para resolver problemas tanto de clasificación (predecir una variable discreta, típicamente binaria) como de regresión (predecir una variable continua). Se trata de modelos excesivamente simples pero, y ahí reside fundamentalmente su interés, fácilmente interpretables.
Existen varios paquetes en R para construir árboles de decisión. De entre todos ellos, vamos a usar los de la librería party:
library(party)Vamos a analizar los datos de Olive.txt, que describen una muestra de diversos aceites de oliva italianos. Para cada aceite se ha anotado su procedencia (Region y Area) y una serie de caracterísicas químicas, las concentraciones de determinados ácidos (como el eicosenoico). Este conjunto de datos se compiló para poder crear un modelo que distinguiese la procedencia del aceite a través de pruebas químicas y evitar así fraudes de reetiquetado. Nuestro objetivo será, por tanto, predecir la zona de procedencia a partir de la huella química de los aceites, es decir, usando esas concentraciones químicas como potenciales predictores del origen de los distintos aceites.
En primer lugar vamos a leer y preparar los datos. Vamos a tratar de predecir la Region, por lo que eliminaremos las variables Area y la auxiliar Test.Training.
olive <- read.table("data/Olive.txt", header = T, sep = "\t")
olive.00 <- olive
olive.00$Area <- olive.00$Test.Training <- NULLA continuación, crearemos el modelo.
modelo <- ctree(Region ~ ., data = olive.00)La fórmula Region ~ . indica que queremos modelar Region en función de ., es decir, el resto de las variables disponibles en nuestros datos. Alternativamente, se pueden indicar las variables predictoras a la derecha de ~ separadas por el signo +: y ~ x1 + x2 + x3. También es posible seleccionar todas menos algunas de ellas con el signo -, p.e., y ~ . - x1. Desafortunadamente, la función ctree no interpreta correctamente expresiones que usan el signo -. Ese es el motivo por el que hemos creado un conjunto de datos auxiliar olive.00; lo natural hubiese sido especificar Region ~ . - Area - Test.Training.
El objeto modelo construido con ctree contiene toda la información relativa al árbol que hemos construido. La mejor manera de inspeccionar el modelo obtenido es representándolo gráficamente haciendo:
plot(modelo)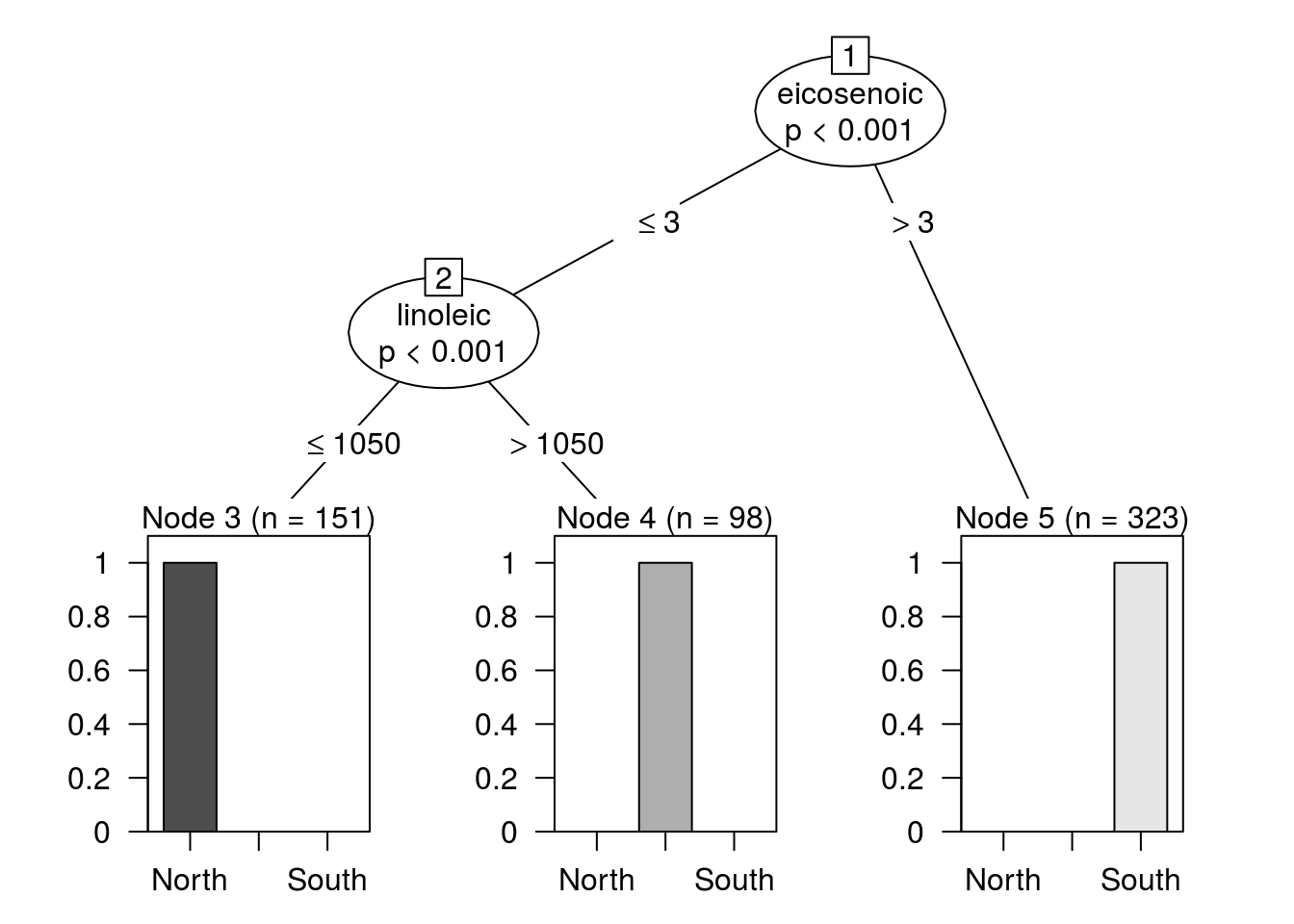
Como puede apreciarse, la predicción es perfecta: en los nodos finales no hay confusión de regiones. Por ejemplo, el nodo terminal de la derecha, el que se construye con la regla eicosenoic > 3, contiene únicamente aceites procedentes de la zona sur de Italia. Por lo tanto, siempre que un aceite cumpla dicha regla, se categorizará como procedente de esa región.
Si en el gráfico resultante no puedes apreciar correctamene los nodos terminales o sus etiquetas, trata de ampliar la ventana gráfica. Si aun ampliándola siguiesen sin verse adecuadamente, guarda el gráfico con una resolución alta en disco y ábrelo con un programa de visualización de imágenes.
Describe las reglas que definen los otros dos nodos terminales y las decisiones que se tomarán con los aceites que caigan en ellos.
Normalmente, para medir el poder predicitivo de un modelo, se utiliza un conjunto de datos para entrenar el modelo y otro distinto para evaluarlo. Nuestro conjunto de datos sugiere realizar la partición de acuerdo con la columna (que suponemos generada al azar) Test.Training. Para ello usaremos la función split:
olive.01 <- olive
olive.01$Area <- NULL
olive.01 <- split(olive.01, olive.01$Test.Training)En el código anterior hemos partido el conjunto de datos en dos trozos en función de la columna Test.Training.
Inspecciona el objeto olive.01.
Ahora usaremos la parte Training para ajustar el modelo:
modelo <- ctree(Region ~ ., data = olive.01$Training)
plot(modelo)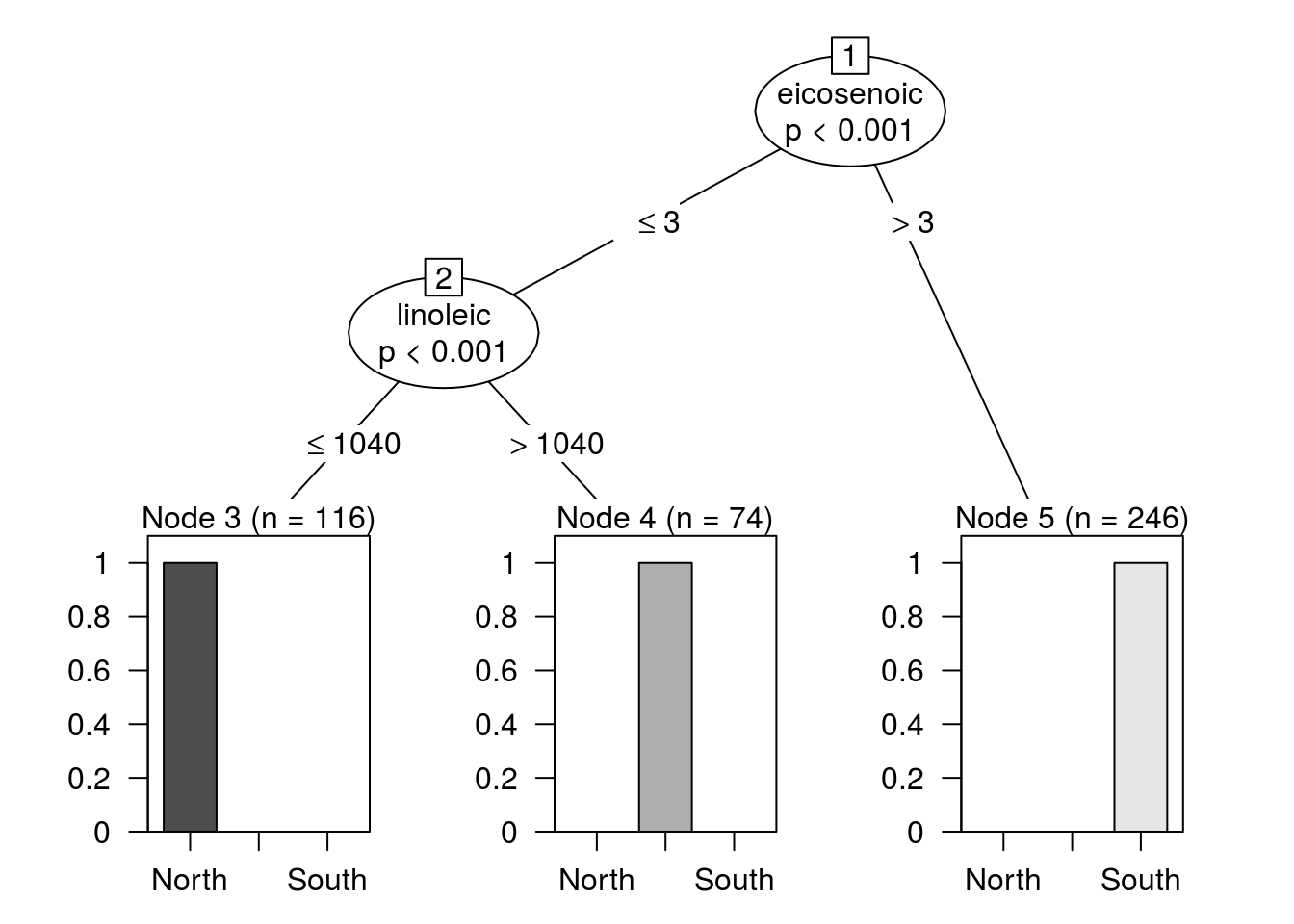
El modelo resultante es muy similar construido con todos los datos. Pero ahora podemos hacer predicciones usando la parte Test34 con la función predict.
predicciones <- predict(modelo, olive.01$Test)La práctica totalidad de los modelos estadíticos en R implementan una versión de predict que siempre admite dos argumentos35: el modelo ajustado previamente y un nuevo conjunto de datos (obviamente, con la misma estructura que el usado para crearlo).
Una vez obtenidas las predicciones, se pueden comparar con los valores originales. La expresión
mean(predicciones == olive.01$Test$Region)## [1] 0.9926471calcula la proporción de aciertos.
¿Por qué?
Cuenta los aciertos y los fallos.
Para ver dónde se han cometido los errores, se puede hacer
table(predicciones, olive.01$Test$Region)##
## predicciones North Sardinia South
## North 34 0 0
## Sardinia 1 24 0
## South 0 0 77que construye una tabla que, idealmente, debería tener una estructura diagonal. Eso correspondería al caso en que cada aceite se asigna a la región a la que realmente pertenece. Sin embargo, el modelo comete un (único) error al asignar a Cerdeña un aceite que, en realidad, es de la región norte.
Repite el ejercicio anterior (predicción) usando la variable Area en lugar de Region. En este caso el modelo no es tan bueno porque hay más áreas que regiones y algunas están tan próximas entre sí que es natural que las diferencias entre sus aceites no sean tan acusadas como entre regiones.
Los bosques aleatorios son más potentes que los árboles que hemos visto arriba. Busca en internet cuál es el paquete y la función que sirve para crear un modelo de bosques aleatorios y repite el ejercicio anterior con él. ¿Funcionan mejor?
El ejercicio anterior sirve de ilustración de cómo en R muchos modelos tienen una interfaz similar. Solo en algunos casos particulares es necesario utilizar sintaxis especiales.